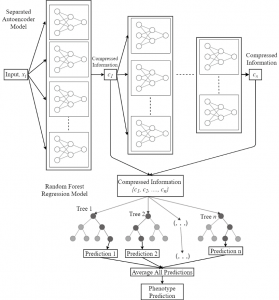
次世代アグリイノベーション研究センター データサイエンスグループ木村彰男准教授らの研究グループは、深層学習を利用したイネの高次元ゲノムデータの圧縮技術、および圧縮後データよりイネ形質の表現型を予測する回帰モデルを開発しました。この成果は学術誌IEEE/ACM Transactions on Computational Biology and Bioinformaticsへの掲載が決定しています。
世界人口の増加が続くなか、持続的かつ安定に食料を増産することは人類の喫緊の課題といえますが、そのためには作物の育種(品種育成)が重要となります。一般的な育種では、交配や選抜を繰り返し、実際の圃場で栽培試験を実施する、というプロセスが必要なため、10年程度の労力が必要、といわれていますが、本研究では、イネのゲノム情報(個体の形質を決定づける遺伝子の総体)を用いた育種に着目しています。ゲノムデータは苗の段階で抽出することも可能なため、育種にかかる時間を短縮させることが期待できますが、ここ数十年の間に明らかにされたゲノムデータは巨大かつ複雑になっており、データ分析のための時間的コストやデータ保存のためのストレージコストが無視できない、という問題も同時に抱えていました。そこで本研究では、この高次元かつ膨大なゲノムデータを、深層学習で広く利用されるオートエンコーダという機構を用いて非常に高い圧縮率でデータ圧縮する技術を開発しました。この方法により、例えば、70万SNP(一塩基多型)からなるHigh-Dimensional Rice Array(HDRA)データセットにおいて、98%程度まで圧縮可能、つまり元のデータセットに対して約2%にまでデータ量を削減することができます。さらに、この小さく圧縮されたゲノム情報から、収量や品質に関連する重要形質の遺伝子型を高い精度で予測するための回帰モデルも開発しており、例えば先のHDRAデータセットなら、最大99%の精度で遺伝子型を予測できることを実証しています。これらの成果により、画期的なイネ品種の育成の加速化や効率化が期待されます。
【発表論文】
タイトル:DeepCGP: A Deep Learning Method to Compress Genome-Wide Polymorphisms for Predicting Phenotype of Rice
著者 :Tanzila Islam, Chyon Hae Kim, Hiroyoshi Iwata, Shimono Hiroyuki, and Akio Kimura
雑誌名 :IEEE/ACM Transactions on Computational Biology and Bioinformatics
DOI番号 :10.1109/TCBB.2022.3231466
( クリックしていただきますと、拡大した画像をご覧いただけます )

![]()
![]()
![]()